首頁 > 技術論壇
R軟體資料探勘實務(下)—集群模型
資料探勘的主要模型可大致分為分類、關聯和集群三大類型。我們已於「R軟體資料探勘實務」(上) (中)兩篇中說明了分類模型和關聯模型,在這裡我們將繼續說明集群模型的應用,並以R軟體的實際操作,找出資料間同質的集群特性。
前言
資料探勘(Data Mining)是透過自動或半自動化的方式對大量的數據進行探索和分析的過程,從其中發掘出有意義或有興趣的現象,進而歸納出有脈絡可循的模式(Model),並藉反覆印證找出意想之外可行的執行方案。
資料探勘的主要模型可大致分為分類(Classification)、關聯(Association)、集群(Clustering)三大類型。我們已於「R軟體資料探勘實務」(上) (中)兩篇中說明了分類模型和關聯模型。因此,在這裡我們將繼續說明集群模型的應用,以R軟體的實際操作,找?資料間同質的集群特性。
一. 鳶尾花資料集的各種集群模型
集群分析是從一大群資料中,獲得知識的基礎過程,目前已廣泛應用在各個領域,包含了資料採礦、統計數據分析…等等。集群分析就是將異質的群體區隔,分成一些同質性較高的子群組或集群,使同一集群內之事務具有高度之相似性(homogeneity),不同集群之事務具有高度之異質性(heterogeneity)。也就是說,不需要事先定義好該如何分類,也不需要訓練組資料,而是靠資料自身的相似性集群在一起,最常使用在市場區隔的應用上。
分群注意事項包括要確保Cluster具代表性,各個Cluster的大小原則上以5-10%為最低門檻,總體Cluster的數量建議為2-12個,過多欄位會產生過多的Cluster,造成分析上的困難。集群分析可分為分割式(Partitional)集群:包括K-Means和K-Medoids clustering等;和非分割式(Non-Partitional)集群:包括Hierarchical和Density-based clustering等。以下我們將使用鳶尾花iris資料集,藉由R軟體的實際操作,來說明集群分析的各種模型:
1. K-平均數(K-Means)集群
K-Means演算法是麥昆(J. B. MacQueen)於1967年正式發表,由於原理簡單、計算快速,很快就成為商用資料採礦軟體中的基本配備。它是屬於前 設式的集群演算法,也就是必須先設定集群的數量,然後根據該設定找出最 佳的集群結構。使同一集群內的資料物件相似性大,不同類別中的資料差異性大,也就是達到「組內同質,組間異質」的處理方式。
K平均數(K-means)是最受使用者歡迎以及最佳的集群分析法之一,屬 分割式分群法,分群方式是先將原始事物分為k個群體,計算某一資料點到集群中心之距離(或相適度),將其分配到最接近的群體,重新計算增加及減少資料點的集群中心,重複計算直至各資料點不必重新分配至其他集群為止。
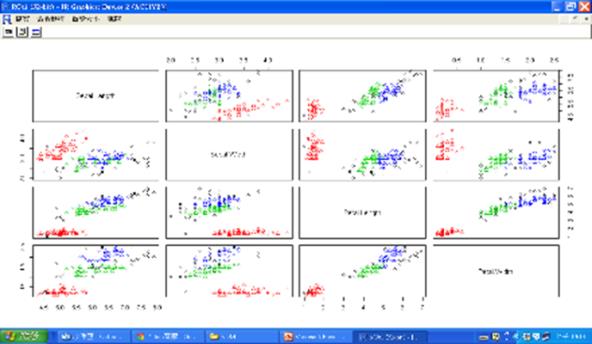
以下我們將藉由R軟體的實際操作,來說明K-Means集群分析。執行 kmeans函數及繪製散佈圖的指令如下:
#iris2=iris[,sapply(iris,is.numeric)]取所有數值資料的指令或者
iris2=iris[,-5]
set.seed(1234)
kmeans.result=kmeans(iris2,3)
kmeans.result
table(iris$Species,kmeans.result$cluster)
plot(iris2,col=kmeans.result$cluster)
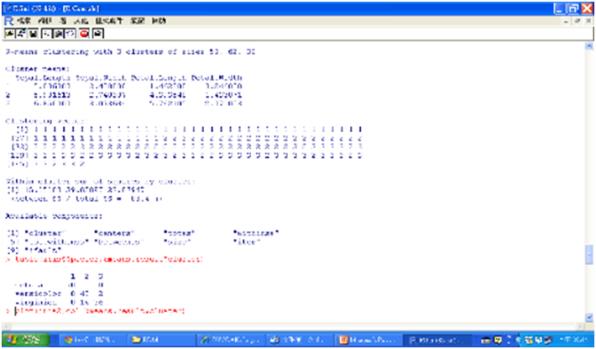
圖一 預測分群結果與找出叢集中心
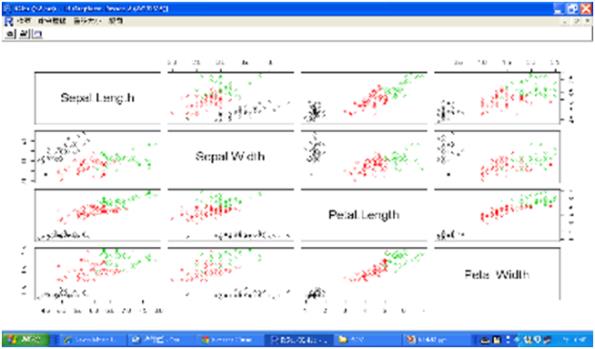
圖二 iris的散佈圖矩陣
接下來我們想利用集群找出離群值並將離群值和集群中心標示出來:
#find 5 largest distances between objects and cluster centers
kmeans.result$centers
centers=kmeans.result$centers[kmeans.result$cluster,]
head(centers)
distances=sqrt(rowSums((iris2-centers)^2))
outliers=order(distances,decreasing=T)[1:5]
outliers
iris2[outliers,]
plot(iris2[c("Sepal.Length", "Sepal.Width")], col=kmeans.result$cluster)
points(kmeans.result$centers[,c("Sepal.Length", "Sepal.Width")], col=1:3, pch=8, cex=2)
points(iris2[outliers,c("Sepal.Length", "Sepal.Width")], col=4, pch='+', cex=2)
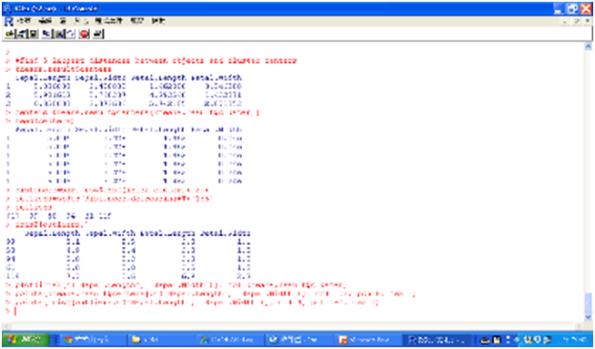
圖三 找出5個離群值
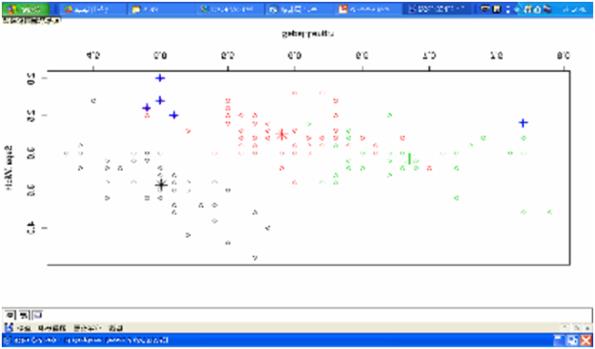
圖四 離群值(+)和集群中心(*)
2. K-物件(K-Medoids)集群
K-物件集群法是以集群中最具代表性的點Medoid作為集群中心,亦即 利用真正的物件代表集群。K-Medoids最經典演算法是分割環繞物件(PAM, Partitioning Around Medoids),由Kaufman and Rousseeuw於1987年提出。 可使用cluster套件的pam函數,或是fpc套件的pamk函數,使用pamk函 數時,使用者不必決定分成幾組,但結果不一定理想。pamk程式碼如下:
iris2=iris[,-5]
library(fpc)
pamk.result=pamk(iris2)
pamk.result
table(iris$Species, pamk.result$pamobject$clustering)
layout(matrix(c(1,2),1,2))
plot(pamk.result$pamobject)
layout(matrix(1))
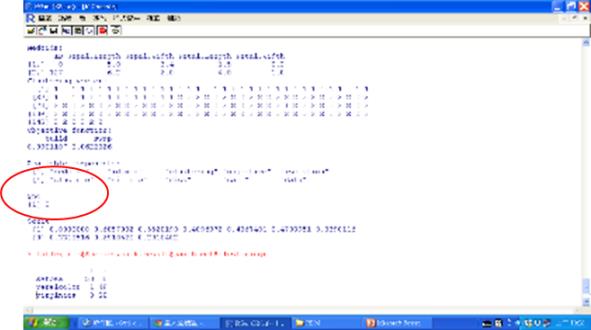
圖五 pamk執行結果(自動分2群)
執行後的pamk集群圖如下,其中Silhouett係數是用來評估集群模型的 準則,計算每筆紀錄(B-A)/max(A,B)的平均值,A是指每筆紀錄到其所屬叢集中心的距離,B是指每筆紀錄到其最近但不屬於的叢集中心的距離。
Silhouett係數範圍在-1(非常差的模型)和1(非常好的模型)之間,圖中的Silhouette係數為(51*0.81+99*0.62)/150=0.69。
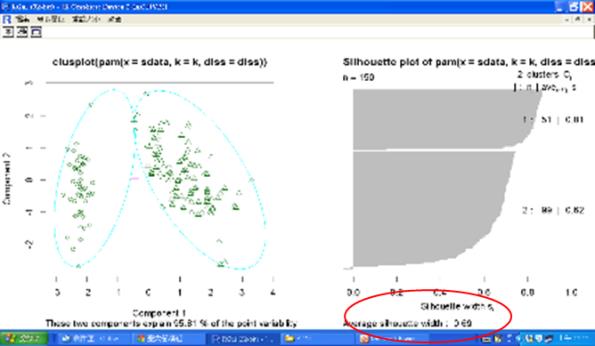
圖六 pamk集群圖
另外我們也亦可使用NbClust套組的NbClust函數,協助使用者決定分群的數目,程式碼如下:
library(NbClust)
iris2=iris[,-5]
result=NbClust(iris2,distance="euclidean",min.nc=2,max.nc=6, method="kmeans", index="all")
Result$Best.partition
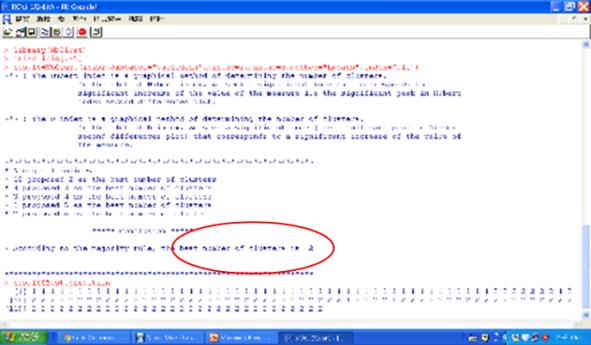
圖七 分群指標執行結果
3. 階層式(Hierarchical)集群
階層式集群是以階層架構的方式反覆進行分裂或聚合,以產生最後的樹狀架構,可從樹狀圖取得任何想要的集群數,缺點是只適合小量資料。常見的階層式集群法包括:
method=”average“,平均距離的平均連鎖法
method=”simple“,最小距離的單一連鎖法
method=”complete“,最大距離的完全連鎖法
method=”ward.D“或 "ward.D2",Ward的最小變異法
由於階層式集群只適合小量資料,在這裡我們只取40筆資料來說明, 程式碼如下:
iris2=iris[,-5]
index=sample(1:nrow(iris2),40) #抽取40筆
irissample=iris2[index,]
#dist函數 distance matrix compute
hclust.result=hclust(dist(irissample),method= “ward.D2”)
hclust.result
plot(hclust.result,labels=iris$Species[index])
rect.hclust(hclust.result,k=3,border="red")
groups=cutree(hclust.result,k=3)
table(iris$Species[index],groups)
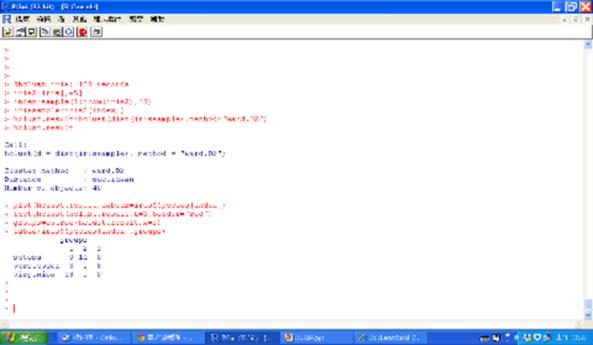
圖八 階層式集群執行結果
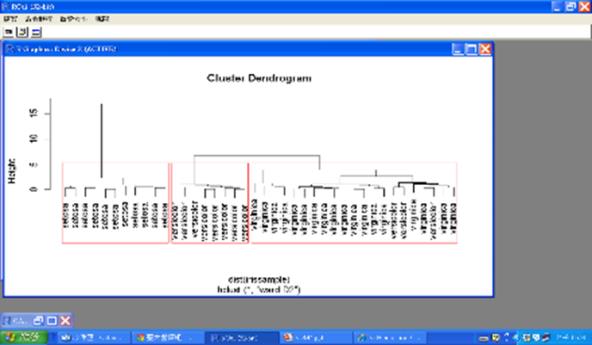
圖九 階層式集群樹狀圖
4. 密度基礎(Density-based)集群
fpc套件的dbscan演算法可提供數值資料的密度基礎集群,dbscan有兩個重要參數,即可達區域eps,定義鄰近地區的大小,以及可達區域的最小點MinPts。如果某個點鄰近地區的點數大於minPts,那麼這個點就是一個密度點,所有從這個點可達的鄰近地區的點就被分成同一個集群。dbscan的程式碼如下:
iris2=iris[,-5]
library(fpc)
dbscan.result=dbscan(iris2,eps=0.42,MinPts=5)
dbscan.result
table(iris$Species, dbscan.result$cluster)
plot(dbscan.result,iris2)
plotcluster(iris2,dbscan.result$cluster)
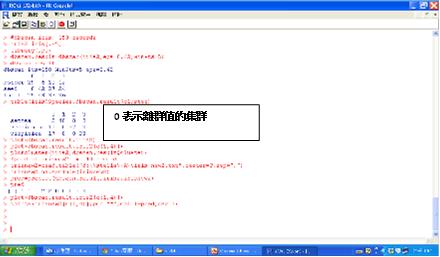
圖十 dbscan的執行結果
圖十一 dbscan集群圖 (黑色是離群值的集群0)
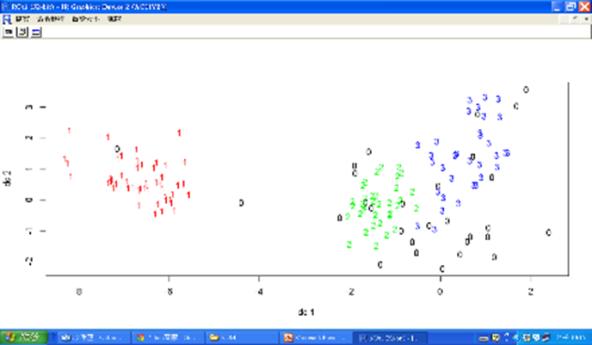
圖十二 0表示離群值的集群
二. 動物園的動物集群範例
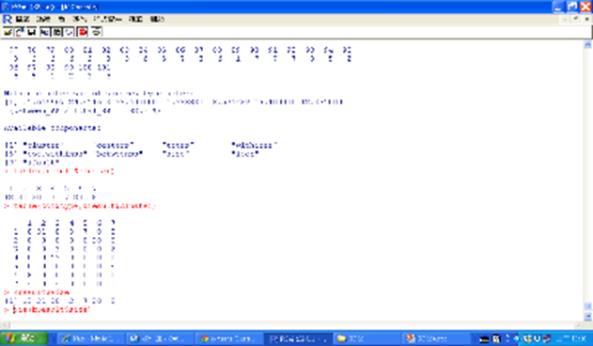
本範例的動物資料集zoo.txt取自美國加州大學歐文分校的機械學習資料庫,將動物分為7群,總共有101筆紀錄,18個欄位說明如下:
1. 動物的名字(animal name):唯一值
2. 是否具有毛髮(hair):Boolean值
3. 身體是否有羽毛(feathers):Boolean值
4. 是否會下蛋(eggs):Boolean值
5. 是否會產奶(milk):Boolean值
6. 是否在空中(airborne):Boolean值
7. 是否在水中(aquatic):Boolean值
8. 是否會獵食(predator):Boolean值
9. 是否有牙齒(toothed):Boolean值
10. 是否有骨幹(backbone):Boolean值
11. 是否會呼吸(breathes):Boolean值
12. 是否有毒(venomous):Boolean值
13. 是否有鰭(fins):Boolean值
14. 腿的數量(legs):{0,2,4,5,6,8}集合的數字
15. 是否有尾巴(tail):Boolean值
16. 是否被馴化(domestic):Boolean值
17. 是否屬貓科(catsize):Boolean值
18. 類型(type):在[1,7]範圍內的整數值
由於本來的資料是將動物分為7種類型,這裡我們也將動物分為7群,並利用混淆矩陣查看分析結果,相關程式碼如下:
zoo=read.table("d:\\stella\\R\\zoo.txt",header=T,sep=",")
zoo=na.exclude(zoo)
#kzoo=zoo[,sapply(zoo,is.numeric)] 取所有數值資料
kzoo=zoo[,-c(1,18)]
set.seed(777)
kresult=kmeans(kzoo,7)
kresult
table(kresult$cluster)
table(zoo$type,kresult$cluster)
kresult$size
pie(kresult$size)
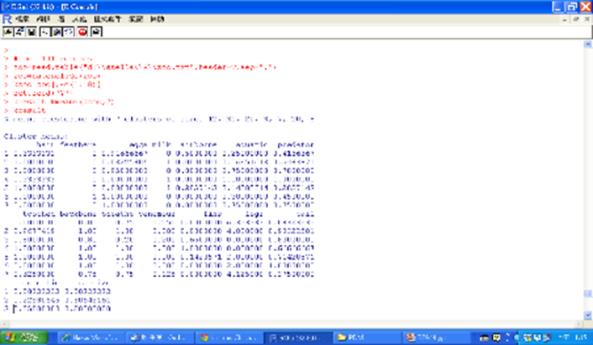
圖十三 預測分群結果與叢集中心
圖十四 混淆矩陣分析的結果
三. 電信公司的客戶集群範例
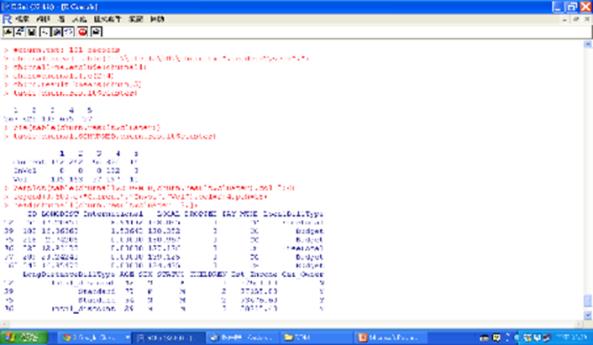
本範例的電信公司的客戶資料集churn.txt總共有1477筆紀錄,15個欄位的說明如下,其中CHURNED欄位將客戶分成Current、Vol、Invol三類:

圖十五
我們將嘗試以LONGDIST、international、LOCAL這三個數值欄位當作輸入,利用kmeans函數建立5個集群的模型,執行的程式碼如下:
churnall=read.table("d:\\stella\\R\\churn.txt",header=T, sep=",")
churnall=na.exclude(churnall)
churn=churnall[,c(2:4)]
churn.result=kmeans(churn,5)
table(churn.result$cluster)
pie(table(churn.result$cluster))
table(churnall$CHURNED,churn.result$cluster)
barplot(table(churnall$CHURNED,churn.result$cluster), col=2:4)
legend(0,600,c("Current","Invol","Vol"),col=2:4,pch=15)
head(churnall[churn.result$cluster==4,]) #Invol都在群集4
圖十六 分群執行結果
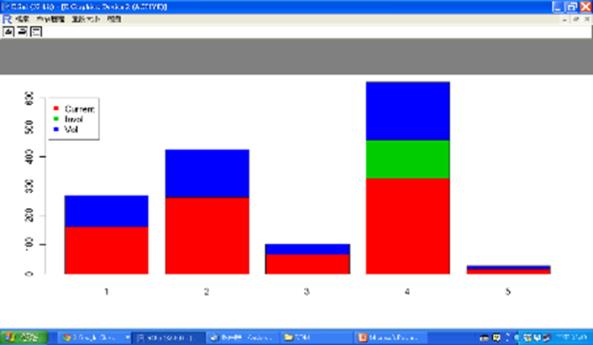
圖十七 CHURNED在叢集中的分配圖
從以上的分析結果,我們發現Invol全部集中在群集4,因此接下來就得進一步探討群集4成員的各種屬性,找出可區別的特性。
四. 蛋白質攝取的集群分析範例
這裡我們將舉一個蛋白質攝取的檔案protein.txt來作說明,共有25個歐洲國家,即25筆紀錄,欄位有country、Redmeat、Whitemeat、Eggs、Milk、Fish、Cereals、Starch、Nuts和Frozen Vegetable,檔案內容如下:
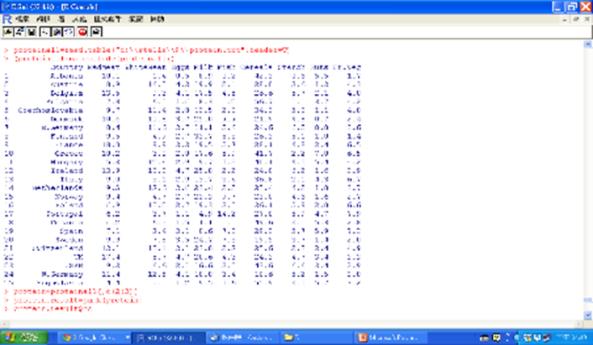
圖十八 蛋白質攝取的檔案
我們將嘗試以紅肉、白肉攝取的這二個數值欄位當作輸入,利用pamk函數找出最佳分組數,再使用kmeans函數建立集群模型,以了解歐洲地區對紅白肉攝取的偏好,執行的程式碼如下:
proteinall=read.table("d:\\stella\\R\\protein.txt",header=T)
(proteinall=na.exclude(proteinall))
protein=proteinall[,c(2:3)]
library(fpc)
protein.result=pamk(protein)
protein.result$nc
protein.result=kmeans(protein,3) #nc
table(protein.result$cluster)
df=data.frame(protein,Country=proteinall$Country, Cluster=protein.result$cluster)
df[order(df$Cluster),]
plot(protein,col=df$Cluster)
以pamk函數找出的最佳分組數為3,於是再使用kmeans函數建立3個集群,之後再根據集群數排序,讓相同集群的國家排在一起,結果如下圖所示:
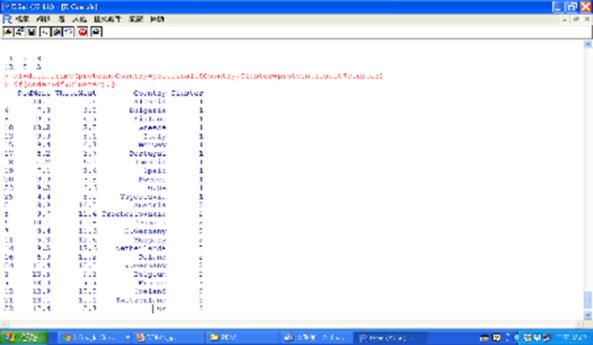
圖十九 蛋白質攝取集群分析結果
不同集群以不同顏色繪製散佈圖,集群1以黑色表示,我們可看出這個集群紅白肉攝取少,配合上圖排序後的結果可知這個集群的國家分佈於北歐和地中海沿岸地區;集群2以紅色表示,我們可看到這個集群白肉攝取多紅肉攝取少,配合上圖排序後的結果可知這個集群的國家大致屬於東歐國家;集群3以綠色表示,我們可看到這個集群紅白肉攝取多,配合上圖排序後的結果可知這個集群的國家大致屬於西歐國家。
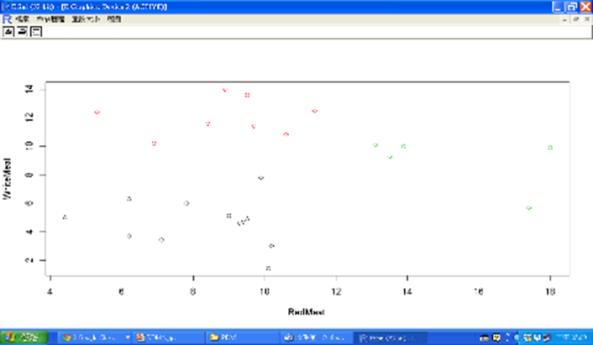
圖二十 蛋白質攝取的集群分析圖