作者:林淑芬 / 臺灣大學計算機及資訊網路中心教學研究組程式設計師
資料探勘對於學術界與實務界而言,是一門兼具問題、理論、與方法的學科。在這裡我們嘗試以不同資料探勘的理論為經,演算方法為緯,在經緯的架構中,藉著不同領域的個案實例,以R軟體的實際操作,說明資料探勘模型所能提供的問題解決方法。
前言
資料探勘(Data Mining)是透過自動或半自動化的方式對大量的數據進行探索和分析的過程,從其中發掘出有意義或有興趣的現象,進而歸納出有脈絡可循的模式(model),並藉反覆印證找出意想之外可行的執行方案。
因此,我們也可以說資料探勘對於學術界與實務界而言,是一門兼具問題、理論、與方法的學科。本文中我們即嘗試以不同資料探勘的理論為經,演算方法為緯,在經緯的架構中,藉著不同領域的個案實例,以R軟體的實際操作,說明資料探勘模型所能提供的問題解決方法。資料探勘的主要模型可大致分為以下三大類:
1. 分類(Classification):分類模型使用一個或多個輸入的值來預測一個或多個輸出目標的值。分類模型可幫助組織預測已知的結果,例如顧客是否購買、流失,腫瘤的判定是良性、惡性。
2. 關聯(Association):關聯模型可找出資料間彼此的相關聯性,其中一個或多個實體與一個或多個其他實體相關聯。關聯模型在預測多個結果時非常有用,例如,購買了產品X的顧客也購買了產品Y和Z。
3. 集群(Clustering):集群模型在不知道特定結果的情況下將數據劃分為具有類似輸入的記錄。在不知道特定結果的情況下,例如想將潛在用戶分成幾個相似的子群組,在客戶群中識別利益群體時,集群模型非常有用。
在這裡我們先用分類的模型舉例,簡易說明如何以R軟體建立和評估模型的實際操作步驟,讓使用者可以輕鬆地了解資料探勘如何幫助我們分析巨量資料,並提供我們潛藏在資料中的有用資訊。
一. 建立分類模型的步驟
本範例檔wdbc.txt是乳腺癌(Breast Cancer Diagnostic)的診斷資料,取自美國加州大學歐文分校的機械學習資料庫,這是Wisconsin大學臨床研究中心於1995年蒐集569例乳腺癌症的病患實際診斷資料,診斷的方式是對於可疑的乳腺腫塊使用細針穿刺的技術 (Fine Needle Aspirate, FNA)蒐集數位化圖像並加以計算,欄位共32項,說明如下:
第1欄:識別號碼(ID number):識別號碼
第2欄:診斷結果(Diagnosis):惡性(M = malignant)、良性(B = benign)
第3-32欄:C1 、C2…C30這30項資料全部都是計算每一個細胞核的真實資料測量值,包含半徑、紋理、周長、範圍、平滑度、緊密度、凹陷部分的程度、凹陷部分的數量、對稱度、碎型維度等。
資料探勘的過程大致分為3個步驟,附上R軟體的指令如下:
1. 讀入資料和資料準備
首先將資料檔wdbc.txt讀到一個框架(data-frame)變數,然後去除遺漏值和不需要的資料(譬如第一欄的資料id),並將資料以70%和30%的比例分割成訓練組(train partition)和測試組(testing partition)。
wdbc=read.table("d:s\\stella\\R\\wdbc.txt", header=T, sep=",")
wdbc=na.exclude(wdbc)
wdbc=wdbc[,-1]
# test.index sampling 30% as testing group, remaining 70% as train group
n=0.3*nrow(wdbc)
test.index=sample(1:nrow(wdbc),n)
wdbc.train=wdbc[-test.index,]
wdbc.test=wdbc[test.index,]
2. 建立模型並輸出決策樹
這個步驟我們要安裝載入所需套件(packages),執行建立模型的函數,輸出決策樹圖形。在這裡我們先試試tree套件的tree函數。
install.packages("tree")
library(tree)
wdbc.tree=tree(diagnosis~.,data=wdbc.train)
wdbc.tree
summary(wdbc.tree)
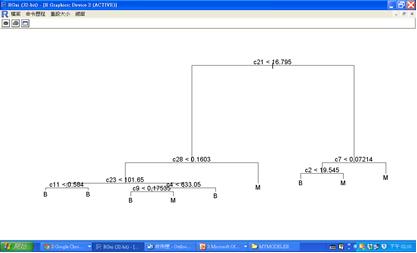
plot(wdbc.tree)
text(wdbc.tree)
3. 結果分析
我們將在這個步驟產生訓練組和測試組的混淆矩陣,以及計算預測的正確率。
#train confusion matrix
diagnosis.train=wdbc$diagnosis[-test.index]
train.pred=predict(wdbc.tree,newdata= wdbc.train, type='class')
(table.train=table(diagnosis.train,train.pred))
cat("Total records(train)=",nrow(wdbc.train), "\n")
cat("Correct Classification Ratio(train)=", sum(diag(table.train))/sum(table.train)*100,"%\n")
#test confusion matrix
diagnosis.test=wdbc$diagnosis[test.index]
test.pred=predict(wdbc.tree,newdata=wdbc.test, type='class')
(table.test=table(diagnosis.test,test.pred))
cat("Total records(test)=",nrow(wdbc.test),"\n")
cat("Correct Classification Ratio(test)=", sum(diag(table.test))/sum(table.test)*100,"%\n")
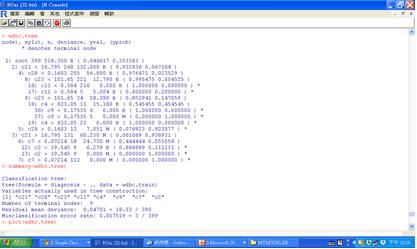
圖一 建立分類模型
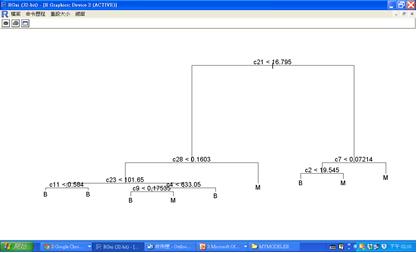
圖二 決策樹輸出
圖三 訓練組和測試組的混淆矩陣以及預測正確率
最後輸出訓練組和測試組的混淆矩陣和正確率分析結果,我們可看到訓練組隨機抽出的7成資料(399筆)中,原本診斷為良性,而使用tree模型,也被分類為良性的有257筆;原本診斷為惡性,而使用tree模型,也被分類為惡性的有139筆,只有3筆分類錯誤,正確率高達99.248%。測試組隨機抽出的3成資料 (170筆)中,原本診斷為良性,而使用tree模型,也被分類為良性的有97筆;原本診斷為惡性,而使用tree模型,也被分類為惡性的有63筆,只有10筆分類錯誤,正確率也高達94.118%。
二. R軟體常用的分類函數
決策樹(decision tree)是常用的資料探勘技術,將資料依照每一階段不同的條件作循環切割(recursive partition),,跟迴歸分析最大的不同在於一個解釋變數可在不同的切割階段被重複使用。決策樹可用於分類預測,此類決策樹稱為分類樹(classification tree),有些決策樹演算法可達成類似迴歸分析的數值應變數預測功能,此類決策樹稱為迴歸樹(regression tree)。
以下我們將介紹幾種R軟體常用的分類函數,記得要先安裝載入套件,才可以執行建立模型的函數:
1. C5.0決策樹(R提供C50套件的C5.0函數)
Quinlan在1986年所提出的ID3演算法後,因其無法處理連續屬性的問題且不適用在處理大的資料集,因此1993又發表C5.0的前身4.5,直到現在所使用的C5.0決策樹演算法。C5.0演算法的結果可產生決策樹及規則集兩種模型,並且依最大資訊增益的欄位來切割樣本,並重複進行切割直到樣本子集不能再被分割為止。C5.0能處理連續型變數與類別型的變數資料,目標欄位必須是類別型變數。這裡我們利用內建的iris資料集來建立模型。
install.packages("C50")
library(C50)
iris.C5=C5.0(Species~ . ,data=iris)
summary(iris.tree)
plot(iris.tree)
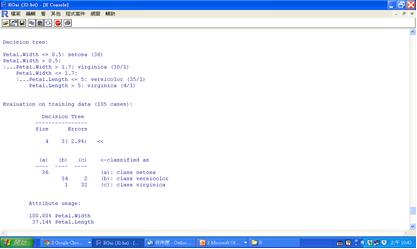
圖四 C5.0規則組及預測變數重要性
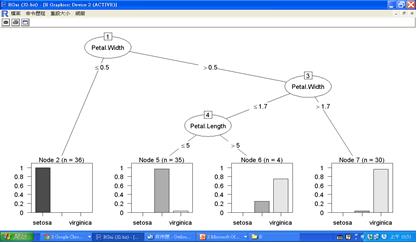
圖五 C5.0的決策樹圖
2. CART分類迴歸樹(R提供tree套件的tree函數或rpart套件的rpart函數)
分類迴歸樹(CART, Classification and Regression Tree)由Brieman在1984年提出CART,以反覆運算的方式,由根部開始反覆建立二元分支樹,直到樹節點中的同質性達到某個標準,或觸發反覆運算終止條件為止。CART的應變數欄位既可以是類別型資料,也可以是數值型資料。以下用到的的範例資料babies.txt,筆數計有1236筆,共有7個欄位,包括bwt(嬰兒體重)、gestation(懷孕日數)、parity(胎序,懷孕過幾胎)、age(母親年齡)、height(母親身高)、weight(母親體重)、smoke(母親抽煙與否,1表抽煙,0表不抽煙)。為了要研究影響嬰兒體重的因素有哪些,這裡使用的是rpart函數,而且預測變數bwt是數值變數。
babies=read.table("d:/stella/R/babies.txt",header=T)
n=0.3*nrow(babies)
test.index=sample(1:nrow(babies),n)
babies.train=babies[-test.index,]
babies.test=babies[test.index,]
install.packages("rpart")
library(rpart)
babies.tree=rpart(bwt~gestation+parity+age+height+ weight+smoke, data= babies.train)
babies.tree
plot(babies.tree)
text(babies.tree)
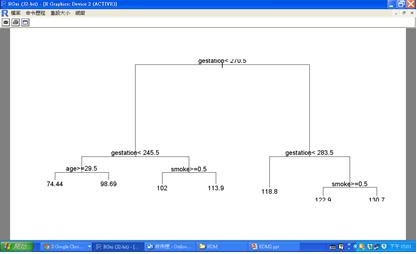
圖六 rpart的決策樹圖
當預測變數是數值變數時,我們無法產生混淆矩陣和預測正確率分析,故使用平均絕對值百分誤MAPE(Mean Absolute Percentage Error)來評估數值變數的預測效果,其公式為各個樣本的(實際值-預測值)取絕對值後除以實際值的平均。當MAPE<10%,則預測效果為很不錯;當10%<MAPE<20%,則預測效果為還好;當MAPE>20%,則預測效果為不佳。我們這個例子的計算結果呈現約是10-11%的MAPE,故預測效果算是不錯。程式碼及執行結果如下:
bwt.train=babies$bwt[-test.index]
train.pred=predict(babies.tree,newdata=babies.train,type="vector")
train.MAPE=mean(abs(bwt.train-train.pred)/bwt.train)
cat("MAPE(train)=",train.MAPE*100,"%\n")
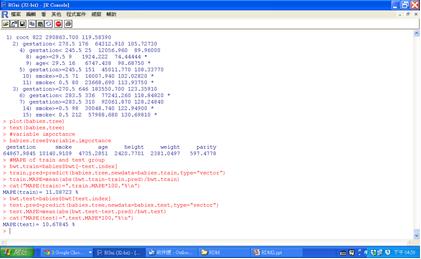
圖七 MAPE的預測效果
3. Random Forest隨機森林(R提供randomForest套件的randomForest函數)
隨機森林是由Brieman在2001年提出的Random Forest決策樹,將訓練樣本所有觀察值作多次抽出放回的隨機取樣,再用這些隨機樣本建構出數百數千棵決策樹,一個新物件會被分到哪個分類是由許多樹共同投票來決定。隨機森林可以應用在分類,也可以用在集群分析的領域。以下是程式碼及iris三個品種的集群分析結果圖:
install.packages("randomForest")
library(randomForest)
#clustering
(iris.clutrf=randomForest(iris[,-5]))
MDSplot(iris.clutrf,iris$Species,palette=rep(1,3), pch= as.numeric(iris$Species))
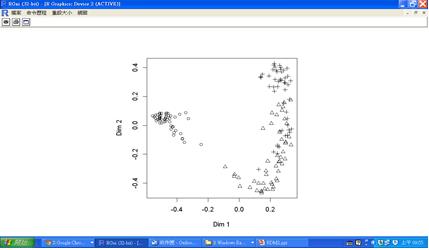
圖八 randomForest集群分析圖
三. 交叉驗證及模型部署
到目前為止我們使用過tree、rpart、C5.0、randomForest等分類函數,但分類正確率的預測又因為是取亂數,每次結果都不盡相同,到底那一個函數比較優越呢?我們希望各做10次分類預測,再取這10次的平均值比較客觀。做法其實很簡單,只要在程式片段前後以迴圈框住即可:
for ( i in 1:10)
{
…
}
下圖即是以隨機森林的randomForest函數交叉驗證10次的執行結果。
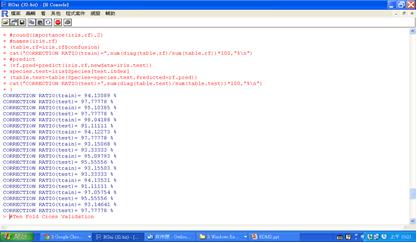
圖九 交叉驗證10次的執行結果
由於我們又將資料分為訓練組和測試組,所以正確率總共有20個數字。以人工運算還是太麻煩,我們再以程式指令將其自動化
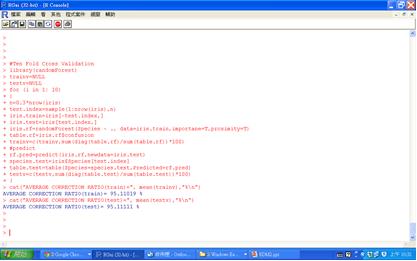
圖十&ensp自動化交叉驗證10次的總結果;
所謂部署(deployment)就是將模型運用在新資料上,亦即先訓練舊資料再以新資料預測。假設新資料檔iris_new2.txt有10筆新資料,有花瓣花萼的長寬,但沒有Species種類欄位需由我們建好的iris模型來進行預測。預測後的分類結果則要和新資料檔合併輸出成一個新檔iris_all.txt。模型部署的程式碼如下:
# irisnew2.txt: 10 records
library(C50)
irisnew2=read.table("d:\\stella\\R\\iris_new2.txt",header=T,sep=",")
#build C5.0 model
iris.tree=C5.0(Species~ . ,data=iris)
#predict irisnew2
iris.pred=predict(iris.tree,irisnew2,type='class')
#merge predict result and output
(irisall=data.frame(irisnew2,Spec.Pred=iris.pred))
write.table(irisall,"d:\\stella\\R\\iris_all.txt",row.names=F)
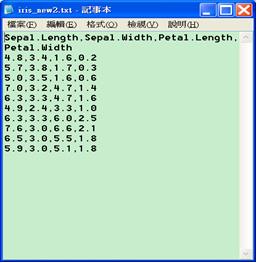
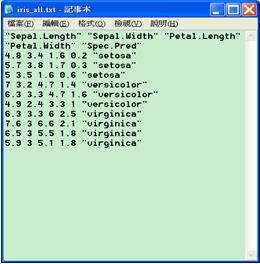
圖十一 新資料檔iris_new2.txt和預測後輸出的新檔iris_all.txt
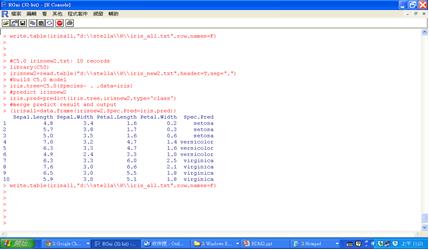
圖十二 預測並輸出合併結果的檔案